TUM verschränkt Grundlagenforschung und Anwendung
2,1 Mio. € zur Erforschung von Mikroorganismen für die Biotechnologie
15.04.2010, Pressemitteilungen
In der mikrobiellen Vielfalt liegt eines der letzten großen Geheimnisse der Biologie: Mikroorganismen leben in unvorstellbarer Menge überall auf der Welt, doch die meisten von ihnen können nicht im Labor kultiviert oder gar genauer untersucht werden. Dabei beinhaltet die unerforschte mikrobielle Biodiversität auch die genetische Basis für neue, biotechnologisch nutzbare Enzyme, Stoffwechselwege und Wirkstoffe. Ein von der Technischen Universität München (TUM) koordinierter Forschungsverbund möchte diesen Schatz jetzt heben und für die industrielle Anwendung in der Biotech-Branche nutzbar machen.
Mikroorganismen sind einfach überall: Sie leben in Luft und Wasser, auf und unter der Erde, bei größter Hitze und in Eiseskälte. Schätzungen zufolge enthält unser Planet bis zu 1000-mal mehr mikrobielle Genome als alle Zellen von Pflanzen und Tieren zusammen. Im Mikroben-Erbgut versteckt sich eine ungeheure genetische Biodiversität in Form von Enzymen, Stoffwechselwegen und Wirkstoffen. Um sie nutzbar zu machen, investiert das Bundesministerium für Bildung und Forschung etwa 2,1 Millionen Euro in den Forschungsverbund ExpresSys (Kurzform für „Neue mikrobielle Expressionssysteme für industriell relevante Gene“): Acht Arbeitsgruppen aus sieben universitären und außeruniversitären Forschungseinrichtungen werden bis 2013 daran arbeiten, die mikrobielle Vielfalt für biotechnologische Anwendungen zu erschließen.
Der Forschungsverbund wird von Prof. Wolfgang Liebl am TUM-Lehrstuhl für Mikrobiologie koordiniert. Die Tücke für die Forscher steckt dabei im Detail. „Leider kann man 99 Prozent der bekannten Mikroorganismen nicht mit den gängigen mikrobiologischen Techniken im Labor züchten“, so Professor Liebl. Mit den klassischen Methoden ist also nur ein geringer Teil der mikrobiellen Schätze der Natur zu heben. Hilfestellung bietet die sogenannte Metagenomanalyse. Mit ihr kann man auch das Erbgut von nicht kultivierbaren Mikroorganismen isolieren und nach biotechnologisch nutzbaren Abschnitten absuchen. Dafür braucht man jedoch so genannte Wirtsorganismen, also gut charakterisierte und für eine gentechnische Modifikation zugängliche Mikroorganismen.
Eine Handvoll Mikroben wird schon seit Jahren gentechnisch verändert und dann in Forschungslaboren wie auch in Fabriken eingesetzt: So werden etwa mit Hilfe des Darmbakteriums Escherichia coli verschiedenste Aminosäuren, Basischemikalien und
pharmazeutische Eiweiße hergestellt, das Bodenbakterium Bacillus licheniformis liefert bestimmte Enzyme, die als Zusatzstoffe für Waschmittel nötig sind. Doch die wenigen etablierten „Bio-Kraftwerke“ sind leider nicht für jede Fragestellung und alle Produktionszwecke einsetzbar. Hier setzt TUM-Professor Liebl mit ExpresSys an: „Wir wollen neue, alternative Wirtsorganismen entwickeln und ihre Nutzbarkeit für biotechnologische Anwendungen ausloten.”
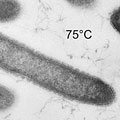
Besonders genau schaut sich der Forschungsverbund dabei einige „Exoten“ an: etwa an extreme Hitze angepasste Bakterien, Mikroorganismen, die Lichtenergie nutzen, sowie Bakterien mit Sozialverhalten. Die Wissenschaftler von TU München, Heinrich-Heine-Universität Düsseldorf, TU Hamburg-Harburg, Universität Hamburg, Universität Duisburg-Essen sowie vom Max-Plack-Institut für terrestrische Mikrobiologie Marburg und vom in Gründung befindlichen Helmholtz-Institut für Pharmazeutische Forschung Saarland hoffen, insbesondere neue Wirtsorganismen zu identifizieren, mit denen man im nächsten Schritt nach Genen für Eiweiß-Biokatalysatoren mit industriellem Anwendungspotenzial suchen kann. Begleitet wird diese Grundlagenforschung von fünf namhaften deutschen Industrieunternehmen. Der kurze Weg in die Anwendung ist also bereits geebnet.
Kontakt:
Prof. Dr. Wolfgang Liebl
Lehrstuhl für Mikrobiologie
Technische Universität München
85350 Freising-Weihenstephan
Tel. 08161 / 71-5450
E-mail: wliebl@wzw.tum.de
http://www.mikro.biologie.tu-muenchen.de
Kontakt: presse@tum.de
Mehr Information
| 100415_PI-ExpresSys.pdf |
Druckversion dieser Pressemitteilung,
(Type: application/pdf,
Größe: 138.4 kB)
Datei speichern
|